文章信息
文章题目:Advancing protein evolution with inverse folding models integrating structural and evolutionary constraints
期刊:Cell
发表时间:2025 年 7 月 7 日
主要内容:中国科学院遗传与发育生物学研究所高彩霞团队在 Cell 杂志发表题为“Advancing protein evolution with inverse folding models integrating structural and evolutionary constraints”的研究论文。该研究基于整合了结构与进化约束的通用逆折叠模型,开发了一种新型人工智能蛋白质工程计算模拟方法 AiCE (AI-informed Constraints for protein Engineering)。该方法无需训练专属人工智能模型,即可实现蛋白质高效进化模拟和功能设计。研究团队利用 AiCE 对多种基因编辑工具进行进化优化,成功实现了其效率和精度的快速提升。
原文链接:
https://www.cell.com/cell/abstract/S0092-8674(25)00680-4
使用TransGen产品:
TransDetect® PCR Mycoplasma Detection Kit (FM311)

背景介绍
蛋白质工程通过人工改变氨基酸序列修饰改造蛋白质结构和功能,相比基因组工程能直接操纵蛋白质分子,借助突变迭代积累快速优化创新蛋白功能,速度远超自然演变,市场潜力巨大。但目前主要改造策略存在依赖经验、实验周期长、成本高的问题,限制规模化应用,而近年来兴起的基于特定蛋白专有 AI 模型的方法,又面临通用性欠佳及模型训练和下游验证成本高的问题,因此有必要开发高效、普适且无需复杂模型训练的蛋白质工程计算模拟策略,以减少计算负荷、实现性能最大化,这对推动蛋白质改造意义重大。
文章概述
蛋白质逆折叠是借助 AI 模型,根据给定三维结构预测可兼容序列的过程,像 ESM-IF1 和 ProteinMPNN 这类通用逆折叠模型,通过对天然蛋白质结构和序列的训练,能隐式学习蛋白质骨架的几何与物理特性,捕捉由进化动力学塑造的蛋白质序列的复杂分布模式。研究团队基于现有通用逆折叠模型开发出 AiCEsingle 模块,具体是依据给定的蛋白质三维结构,对逆折叠模型输出的氨基酸序列进行采样,提名高频出现的氨基酸类型,再通过结构约束对氨基酸频率进行差异筛选,得到最终预测的单个氨基酸替换类型。团队用 60 个深度突变扫描(DMS)数据测试该模块性能,发现其预测准确率达 16%;通过消融实验和逻辑回归分析,证实结构限制在该方法中的必要性,相比无限制方案性能提升 37%;进一步平行比较分析显示,其性能较其他常见 AI 模型提升 36%-90% 以上。从蛋白类型而言,AiCEsingle 能有效进化复杂蛋白和蛋白质-核酸复合物(如 CRISPR 蛋白、SARS-CoV-2 病毒蛋白等),通用性广泛。为克服突变组合中常见的负向上位效应,研究团队假设存在进化耦合的氨基酸位置可能具有功能协同性,进而构建了通过预测进化耦合性来确定突变组合位置的 AiCEmulti 模块。对 6 个突变文库的分析结果表明,AiCEmulti 与蛋白质大模型 SaProt 的预测能力相当,但计算成本很低。团队建立的包含这两类模块的 AiCE 方法,可快速有效预测单突和组合突变,该方法利用现有通用逆折叠模型,无需重新或迁移训练专有蛋白模型,大大降低了计算成本,仅需 1.15 个 CPU 时就能识别 SpCas9 蛋白(>1000 个氨基酸)的单突和双突变体。
利用这一方法,研究团队进一步在湿实验层面完成了对脱氨酶、核定位序列、核酸酶和逆转录酶等 8 种结构与功能各异的蛋白质的 AiCE 功能验证,由此证明了该方法具有简单、高效且通用的特点。依托优化后的脱氨酶,团队深入研发出可应用于精准医疗和分子育种的新型碱基编辑器,其中包括编辑窗口缩小近一半的新型胞嘧啶碱基编辑器 enABE8e、保真度提升 1.3 倍的新型腺嘌呤碱基编辑器 enSdd6-CBE,以及活性提升 13 倍的新型线粒体碱基编辑器 enDdd1-DdCBE。
综上所述,这项研究开发了一种基于人工智能的新型蛋白质工程计算模拟方法 AiCE。与传统蛋白质工程方案相比,该方法在效率、可扩展性和通用性方面均展现出显著优越。通过计算模拟甚至替代湿实验,是当前生命科学领域的重要发展趋势和前沿方向,而本研究在此方面开展的探索具有积极意义。当前,基于人工智能的蛋白质分析工具往往依赖大量计算资源,这对许多实验室而言难以获取。而本项工作表明,通过开发更高效的生物信息学工具,能够最大限度降低计算负荷,从而让更多生物学家切实享受到人工智能技术带来的科研便利。
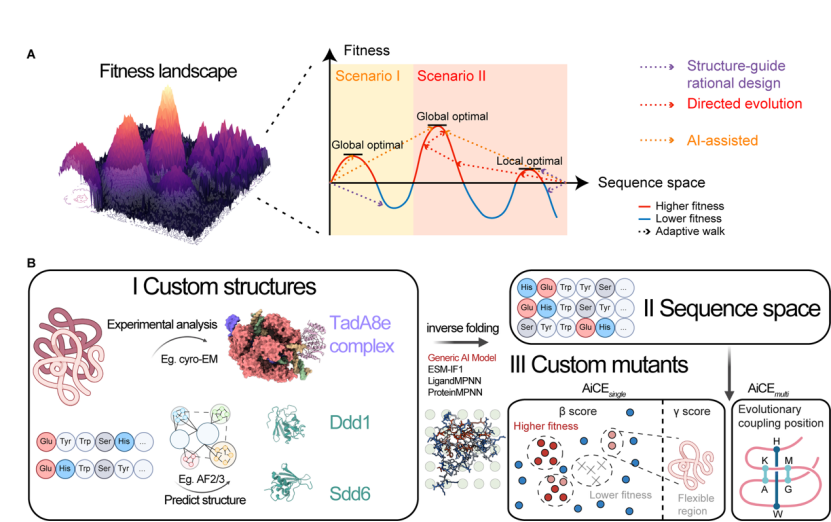
常见蛋白质工程方法的示意图和 AiCE 方法概述
乐动平台(中国)生物产品支撑
优质的试剂是科学研究的利器。乐动平台(中国)生物的支原体检测试剂盒(PCR法)(FM311)助力本研究。产品自上市以来,深受客户青睐,多次荣登知名期刊,助力科学研究。
TransDetect® PCR Mycoplasma Detection Kit (FM311)
一款通过 PCR 方法检测培养细胞等生物材料中的支原体,针对支原体 16S rRNA 序列保守区域设计特异引物,直接使用细胞培养液作为模板,特异性扩增支原体 DNA。
产品特点
• 检测灵敏度高、准确性好。
• 特异性强,只扩增支原体 DNA。
• 操作简便,无需提取基因组 DNA。
乐动平台(中国)生物的产品再度亮相 Cell 期刊,不仅是对乐动平台(中国)生物产品卓越品质与雄厚实力的有力见证,更是生动展现了乐动平台(中国)生物长期秉持的“品质高于一切,精品服务客户”核心理念。一直以来,乐动平台(中国)生物凭借对品质的执着追求和对创新的不懈探索,其产品已成为众多科研工作者信赖的得力助手。展望未来,我们将持续推出更多优质产品,期望携手更多科研领域的杰出人才,共同攀登科学高峰,书写科研创新的辉煌篇章。
使用 TransDetect® PCR Mycoplasma Detection Kit (FM311) 产品发表的部分文章:
• Liu J L, Yan X Q, Wu H, et al. RNA codon expansion via programmable pseudo uridine editing and decoding[J]. Nature, 2025.(IF 50.5)
• Fei H Y, LI Y J,Liu Y J, et al. Advancing protein evolution with inverse folding models integrating structural and evolutionary constraints[J]. Cell, 2025.(IF 45.6)
• Nian Z, Dou Y, Shen Y, et al. Interleukin-34-orchestrated tumor-associated macrophage reprogramming is required for tumor immune escape driven by p53 inactivation[J]. Immunity, 2024.(IF 25.5)
• Xu J, Liang Y, Li N, et al. Clathrin-associated carriers enable recycling through a kiss-and-run mechanism[J]. Nature Cell Biology, 2024.(IF 17.3)
• Wang J, An Z, Wu Z, et al. Spatial organization of PI3K-PI (3, 4, 5) P3-AKT signaling by focal adhesions[J]. Molecular Cell, 2024.(IF 14.5)
• Nian Z, Zheng X, Dou Y, et al. Rapamycin pretreatment rescues the bone marrow AML cell elimination capacity of CAR-T cells[J]. Clinical Cancer Research, 2021.(IF 11.4)
• Xu D, Zhao H, Jin M, et al. Modulating TRADD to restore cellular homeostasis and inhibit apoptosis[J]. Nature, 2020.(IF 50.5)